Einleitung
Influenza
A- und B-Viren gehören zu den wichtigsten Pathogenen des
Menschen. Influenzavirus-Infektionen treten regelmäßig
in den Wintermonaten in epidemischer Form auf und verursachen
hoch fieberhafte Erkrankungen bei ca. 20% der Kinder und 5%
der Erwachsenen [1, 2].
Ein
charakteristisches Merkmal der Influenzaviren ist, dass sie
starken Veränderungen unterworfen sind, von denen vor allem
die beiden an der Virusoberfläche gelegenen Antigene, das
Hämagglutinin (HA) und die Neuraminidase (NA) betroffen
sind. Kontinuierliche Veränderungen werden als Antigen-Drift
bezeichnet und kommen sowohl bei Influenza A- als auch bei Influenza
B-Viren vor. Diese Antigen-Drift ist die Ursache dafür,
dass Influenzavirus-Infektionen keine lang anhaltende Immunität
hinterlassen und, dass es daher immer wieder zu Reinfektionen
und jährlichen Epidemien kommt. Plötzliche und drastische
Veränderungen, die als Antigen-Shift bezeichnet werden,
konnten bis jetzt nur bei Influenza A-Viren beobachtet werden
und treten in unvorhersehbaren Intervallen auf. Wenn solche
so drastisch veränderte Virusvarianten die Fähigkeit
besitzen effizient von Mensch zu Mensch übertragen zu werden,
kann es zu ausgedehnten Epidemien und in weiterer Folge zu einer
Pandemie kommen. Dies geschah nach dem derzeitigen Kenntnisstand
in der Geschichte des Influenzavirus bisher zweimal und verursachten
die Pandemien von 1958 und 1968.
Die
starke genetische Variabilität der Influenzaviren stellt
vor allem für die Produktion effizienter Impfstoffe ein
erhebliches Problem dar [3-11]. Da die Wirksamkeit der Influenzaimpfung
in erster Linie von der Ähnlichkeit der in den Impfstoffen
enthaltenen Stämme mit den tatsächlich zirkulierenden
Viren abhängt, muss die Impfstoffzusammensetzung jährlich
neu angepasst werden [12-14].
Aus
diesem Grund hat die Weltgesundheitsorganisation (WHO) ein weltweites
Netzwerk von Referenzlaboratorien für die Charakterisierung
zirkulierender Influenza-Stämme etabliert und basierend
auf den Daten dieses Überwachungssystems wird die Empfehlung
betreffend die aktuelle Impfstoffzusammensetzung für die
nördliche Hemisphäre jährlich im Februar abgegeben.
Um
die Aufgaben eines Referenzlabors der WHO in Österreich
erfüllen zu können, hat das Institut für Virologie
der Medizinischen Universität Wien unterstützt von
Roche-Austria das Diagnostische Influenza Netzwerk Österreich
(DINÖ) in der Wintersaison 2000/2001 ins Leben gerufen.
Das DINÖ ist ein Sentinella-System, an dem wenige ausgewählte
niedergelassene Ärzte in allen Bundesländern mitarbeiten
und Proben (Nasen-Rachenabstriche) von selektionierten Patienten
mit akuten Influenza-ähnlichen Symptomen einsenden. Die
labordiagnostische Absicherung der klinischen Verdachtsdiagnose
Influenza erfolgt durch den Nachweis von Influenzaviren in den
Nasen-Rachenabstrichen mittels molekularbiologischer (RT-PCR)
Methoden und der Virusisolierung in Zellkultur. Art und Ausmaß
der Veränderung der zirkulierenden Virusstämme wird
durch antigene und genetische Charakterisierung ihrer Oberflächenglykoproteine
bestimmt [15].
Die
im Rahmen des DINÖ eingesendeten und untersuchten Proben
repräsentieren eine Stichprobe, die österreichweit
das Auftreten der Influenza-Viren erfasst und deren genaue Typisierung
ermöglicht. Durch die Analyse dieser Daten kann eine rasche
Aussage über die Wirksamkeit der für die jeweilige
Saison verfügbaren Impfstoffe gemacht werden.
Abbildung
1: Neuerkrankungen an Grippe/grippalem Infekt und die
Gesamtanzahl an positiven Virusnachweisen pro Woche während
der 4 analysierten Saisonen
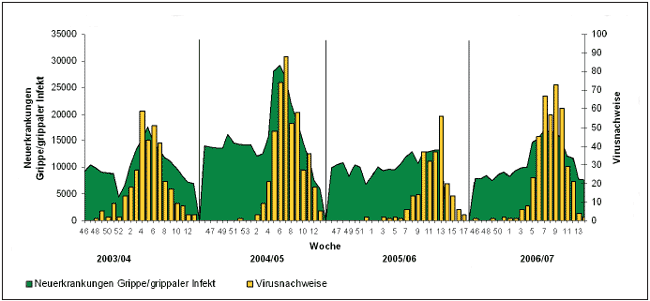
Abbildung
1 gibt einen Überblick über die Daten von 4 Influenza-Saisonen
(2003/04 bis 2006/07) in Österreich. Die Auswertung der
virologischen Ergebnisse gemeinsam mit den epidemiologischen
Daten der Magistratsabteilung 15 der Stadt Wien ermöglichen
den Beginn, das Ausmaß und das Ende der alljährlichen
Grippewelle sehr genau zu erfassen. In allen untersuchten Saisonen
konnte bereits einige Wochen vor dem für Influenzawellen
charakteristischen signifikanten Anstieg der Erkrankungszahlen
an Grippe/grippalem Infekt sporadische Fälle von Influenzavirus-Infektionen
nachgewiesen werden.
Die
jeweils dominierenden Stämme, die neu aufgetretenen Drift-Varianten
und die Übereinstimmung der zirkulierenden Stämme
mit den für die entsprechende Saison empfohlenen Impfstämmen
sind in Tabelle 1 zusammengefasst.
Tabelle
1: Gegenüberstellung der zirkulierenden Influenzaviren
zu den verwendeten Impfstämmen der jeweiligen Grippesaison
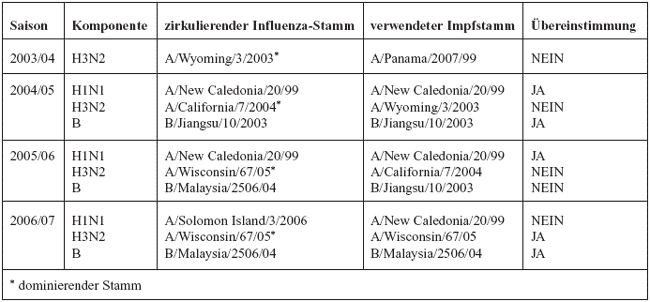
In
der Saison 2003/04 startete die Influenzavirusaktivität
in Kalenderwoche 2 und diese Grippewelle kann sowohl was ihre
Dauer von 9 Wochen, als auch das Ausmaß und die Schwere
der Erkrankungen betrifft, als durchschnittlich bezeichnet werden.
Der Höhepunkt der Neuerkrankungen wurde in den Kalenderwochen
4 bis 6 verzeichnet, wobei alle Influenzaerkrankungen in dieser
Saison durch den Subtyp A/H3N2 verursacht wurden. Die genaue
Stammanalyse ergab, dass überwiegend A/H3N2-A/Wyoming/3/03-ähnliche
Stämme zirkulierten. Im Impfstoff für diese Saison
war jedoch A/Panama/2007/99 (H3N2) enthalten, wodurch die Impfung
keinen optimalen Schutz gegen die tatsächlich zirkulierenden
Stämme bieten konnte. Darüber hinaus zeigte die genaue
Genotypisierung das zusätzliche Auftreten von neuen A/H3N2-Drift-Varianten,
die bereits eine große Ähnlichkeit mit dem in der
folgenden Saison zirkulierenden A/H3N2-Stamm aufwiesen.
Diese
folgende Saison (04/05) war für Virologen besonders interessant,
da beide Typen, mehrere Subtypen und Stämme gleichzeitig
zirkulierten, wobei zwei Drittel der Erkrankungen durch Influenza
A- und ein Drittel durch Influenza B-Viren verursacht wurden.
Zusätzlich konnte bei den Influenza A-Viren auch eine Ko-Zirkulation
der beiden Subtypen A/H3N2 (67%) und A/H1N1 (33%) nachgewiesen
werden. Wenn man bedenkt, dass in der Regel nur ein Stamm eines
Subtyps dominiert, ist es nicht weiter verwunderlich, dass während
dieser Grippewelle bis zu 30 000 Neuerkrankungen an Grippe/grippalem
Infekt pro Woche zu verzeichnen waren.
Die
Daten der Stammcharakterisierungen ergaben, dass die zirkulierenden
Influenza A/H1N1-Viren (A/New Caledonia/20/99-like) und Influenza
B-Viren (B/Jiangsu/10/2003-like) den in den Impfstoffen für
diese Saison enthaltenen Impfstämmen entsprachen, dass
jedoch die A/H3N2-Komponente der Impfstoffe (A/Wyoming/3/03)
nicht ideal mit den tatsächlich zirkulierenden A/H3N2 A/California/7/2004-ähnlichen
Viren übereinstimmte. Auch in dieser Saison konnte wieder
das Neuauftreten einer A/H3N2-Drift-Variante (A/Wisconsin/67/05)
nachgewiesen werden, die dann den Großteil der Influenzavirus-Infektionen
der darauffolgenden Saison verursachte. Diese (2005/06) war
durch ihren späten Beginn in der Kalenderwoche 7 und eine
ungewöhnlich lange Dauer von ca. 10 Wochen gekennzeichnet.
Besonders auffällig war dabei jedoch, dass der epidemische
Schwellenwert von 10000 – 12000 Neuerkrankungen an Grippe/grippalem
Infekt pro Woche nur marginal überschritten wurde. Wie
im Jahr zuvor, konnte auch in dieser Saison die Ko-Zirkulation
von Influenza A-(73%) und Influenza B-Viren (27%) beobachtet
werden. Trotz der klinisch moderaten Situation konnten dennoch
mittels der laufend durchgeführten Genotypisierungen vier
verschiedene zirkulierende Varianten des Influenza A-Virus identifiziert
werden. Zwei davon (A/NewCaledonia/20/99 (H1N1) und A/California/7/2004
(H3N2)) entsprachen den für diese Saison konzipierten Impfstämmen,
wobei letzterer gemeinsam mit dem bereits in der Vorsaison als
Drift-Variante aufgetretenen A/Wisconsin/67/05(H3N2) die dominierenden
Stämme dieser Saison waren. Zusätzlich konnte noch
im Verlauf dieser Grippewelle eine neu aufgetretene A/H3N2-Drift-Variante
als vierter zirkulierender Influenza A-Stamm identifiziert werden.
Die
Stammcharakterisierung der Influenza B-Viren ergab hingegen,
dass von Beginn an ein neuer Influenza B-Virusstamm (B/Malaysia/2506/04)
zirkulierte, der sich von dem im Impfstoff enthaltenen (B/Shanghai/361/2002)
signifikant unterschied.
Die
Influenzasaison 2006/07 startete in der Kalenderwoche 5 und
dauerte ca. 8 Wochen. Sie wurde von Influenza A/H3N2-Viren dominiert
und es konnten nur einige wenige Influenza A/H1N1- und Influenza
B-Viren nachgewiesen werden. Die genaue genetische Analyse der
zirkulierenden Stämme ergab, dass der Großteil der
Erkrankungen von Influenza A/Wisconsin/67/05-ähnlichen
(H3N2) Viren verursacht worden war, die den in den Impfstoffen
für diese Saison enthaltenen Influenza A/H3N2-Stamm sehr
ähnlich waren. Zusätzlich konnte auch die bereits
in der Vorsaison aufgetretene A/H3N2-Drift-Variante (A/Brisbane/10/07)
wieder nachgewiesen werden. Die nur sehr sporadisch vorkommenden
A/H1N1-Erkrankungen wurden durch eine Drift-Variante des H1N1-Virus
verursacht (A/Solomon Island/3/2006-like), die durch den im
Impfstoff enthaltenen Stamm A/NewCaledonia/20/99 nicht abgedeckt
war.
Die
Daten dieser vier Saisonen zeigen sehr deutlich die komplexe
Dynamik der sich kontinuierlich verändernden Influenzaviren,
die nur durch den Einsatz hochsensitiver antigener und genetischer
Charakterisierungsmethoden erfasst werden kann und unterstreichen
die Bedeutung der virologischen Überwachung für die
Entwicklung möglichst wirksamer Impfstoffe.
|