| Serotypisierung
und molekulare Analyse von Streptococcus pneumoniae-Isolaten aus Österreich |
| U. Straschil 1, A.
Buxbaum 2, W. Graninger 1, A. Georgopoulos 1 1 Universitätsklinik für Innere Medizin I, Abteilung für Infektionen und Chemotherapie, AKH Wien (Leiter: Univ.-Prof. DDr. W. Graninger) 2 Universitätsklinik für Innere Medizin IV, Abteilung für Pulmologie, AKH Wien (Vorstand: Univ.-Prof. Dr. L.H. Block) |
| Schlüsselwörter:
Streptococcus pneumoniae, serotypisieren, molekulare Analyse |
Zusammenfassung Infektionen mit Streptococccus pneumoniae können sowohl bei Kindern als auch Erwachsenen zu schweren Infektionen führen. Im Zuge einer laufenden Pneumokokkenstudie wurden 195 Pneumokokken serotypisiert und davon 44 Stämme mittels Pulsfeldgelelektrophorese (PFGE), AP-PCR und rep-PCR analysiert. Um mögliche genotypische Verwandtschaften aufzuzeigen, wurden auch Stämme aus Ungarn, Deutschland, Frankreich und Spanien untersucht. Der am häufigsten gefundene Serotyp war 19, gefolgt von 23 und 6. Österreichische Penicillin-resistente S. pneumoniae-Stämme vom Serotyp 23 hatten eine große Ähnlichkeit mit dem spanischen und französischen 23F-Klon, aber nicht mit dem deutschen 23F-Klon. Weiters zeigte der österreichische Serotyp 19 und 19A keine genetischen Ähnlichkeiten mit dem ungarischen 19- und 19A-Klon. Das könnte der Hinweis auf einen rein österreichischen Penicillin-resistenten Pneumokokken-Klon sein, der den Serotyp 19 aufweist. Große Unterschiede wurden bei den Penicillin-sensitiven Pneumokokken gefunden. Bei der Typisierung der Stämme erwies sich die PFGE der AP-PCR und der rep-PCR überlegen. |
Key-words: Streptococcus pneumoniae, serotyping, molecular analysis |
Summary Infections due to Streptococcus pneumoniae continue to cause significant morbidity and mortality in both adults and children. As part of an ongoing surveillance program of S. pneumoniae in Austria 195 pneumococci were serotyped and 44 strains were further analysed by pulsed field electrophoresis, arbitrarily primed PCR (AP-PCR) and repetitive PCR (rep-PCR). In order to establish possible genotypic relatedness also strains from Hungary, Germany, France and Spain were examined. The most common serotype found was 19, followed by 23 and 6. Austrian serotype 23 penicillin-resistant S. pneumoniae strains showed close relatedness to both the Spanish and the French 23F clone, but not to the German 23F clone. The fact that the Austrian serotype 19 and 19A PRSP did not share genetic background with the Hungarian 19 and 19A clone, points to the existence of an Austrian PRSP clone expressing serotype 19. Great diversity was found among penicillin-sensitive pneumococci. PFGE when compared to AP-PCR and rep-PCR provided the best intraspecies discrimination. |
Einleitung S. pneumoniae ist eine sehr häufige Ursache für Pneumonien, Bakteriämien, Menigitis und Otitis media [13]. Vor allem ältere oder durch Krankheit bereits geschwächte Personen sind sehr gefährdet. In den letzten 3 Jahrzehnten konnte ein starker Anstieg der Resistenz von S. pneumoniae-Isolaten gegenüber sowohl Betalaktamantibiotika als auch Nicht-Betalaktamantibiotika in vielen Teilen der Welt festgestellt werden [1, 23, 25]. Resistenzraten von Pneumokokken gegenüber Penicillin, die in einem Bereich zwischen 44%-59% liegen, wurden z. B. aus Südafrika und verschiedenen europäischen Ländern berichtet [12, 18]. Die Verteilung der Serotypen variiert je nach Bevölkerung und Land sehr stark. Die unterschiedlichen Serotypen zeigen außerdem unterschiedliche Virulenz. Bis heute wurden mehr als 90 verschiedene Serotypen identifiziert [6]. Die Verteilung der Serotypen zu kennen ist vor allem bei der Entwicklung eines Impfstoffes von besonderer Wichtigkeit [14, 24]. Neben der Serotypisierung gibt es die verschiedensten molekularen Techniken, wie Multilocus-Enzym-Elektrophorese, Profilanalyse von Penicillin-bindenden Proteinen, Ribotypisierung, Pulsfeldgelelektrophorese oder die verschiedensten Arten der PCR [9, 11]. In dieser Studie wurde die Serotypenverteilung von Streptococcus pneumoniae in Österreich, die genetische Verwandtschaft zwischen den einzelnen Serotypen und die Beziehung zwischen Penicillin-resistenten und Penicillin-sensiblen Pneumokokken desselben Serotypes untersucht. Um diese Beziehungs- und Verwandtschaftsverhältnisse untersuchen zu können, wurden die bereits oben erwähnten molekularen Techniken benutzt. |
|
151 Penicillin-empfindliche Streptococcus pneumoniae (PSSP) und 44 Penicillin-resistente Streptococcus pneumoniae (PRSP), die in einem Zeitraum von 2 Jahren in 25 österreichischen Zentren gesammelt wurden, wurden untersucht. Die gesammelten Isolate wurden aus unterem und oberem Respirationstrakt, Auge, Ohr, Blut und Liquor isoliert. Alle 195 PSSP und PRSP wurden serotypisiert. Zur molekularen Analyse wurden 21 österreichische PRSP, 10 österreichische PSSP, 10 PRSP aus Ungarn, Spanien, Frankreich, Deutschland und ein ATCC S. pneumoniae-Stamm (ATCC 49619) herangezogen. Diese Stämme hatten die Serotypen 19, 19A, 23, 23B und 23F. Die Identifikation der Pneumokokken erfolgte nach Standard-Labormethoden. Zur Empfindlichkeitsprüfung wurde eine standardisierte Mikrodilutionsmethode nach NCCLS verwendet [20]. Die verwendeten Sera stammten vom Statens Serum Institut in Kopenhagen, Dänemark. Die PFGE wurde nach einer Methode, wie sie von Mathuschek und Mitarbeitern beschrieben wurde [19], durchgeführt. Die AP-PCR und die rep-PCR wurden nach einer modifizierten Methode, beschrieben von Hermanns und Mitarbeitern, durchgeführt. Die DNA-Analyse erfolgte mittels einer dafür entwickelten Software (Molecular Fingerprinting Plus). |
|
In Tabelle 1 wird die Verteilung der Serotypen nach Penicillin-MHK gezeigt. Tabelle 2 zeigt die Serotypenverteilung in den unterschiedlichen Altersgruppen. Der häufigste Serotyp war 19, gefolgt von 23 und 6. Unter den intermediär-empfindlichen und den resistenten Pneumokokken wurde der Serotyp 23 am häufigsten gefunden. In allen Altersgruppen war die Serotypenverteilung sehr ähnlich, mit Ausnahme der Gruppe > 65, bei der kein Serotyp 19 gefunden werden konnte.
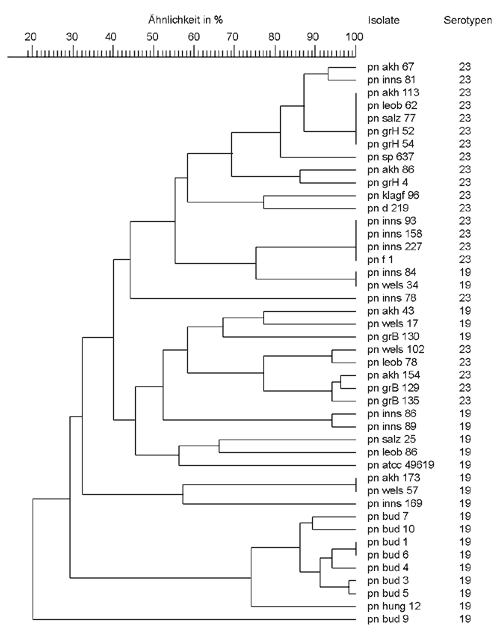
44 Pneumokokkenstämme wurden mittels PFGE analysiert (Tabelle 3). Sowohl bei resistenten als auch bei sensiblen Stämmen desselben Serotypes konnte Polymorphie festgestellt werden. Von 44 Stämmen waren 41 typisierbar. Die Ergebnisse sind in Abbildung 1 dargestellt. Die Ähnlichkeit der DNA-Fragmentmuster bewegte sich in einem Bereich von 44%-100%. Gefunden wurden außerdem drei Clusters mit je zwei Isolaten. Isolate aus Ungarn zeigten keine Cluster, waren aber auch nicht verwandt mit österreichischen, französischen, deutschen oder spanischen Isolaten.
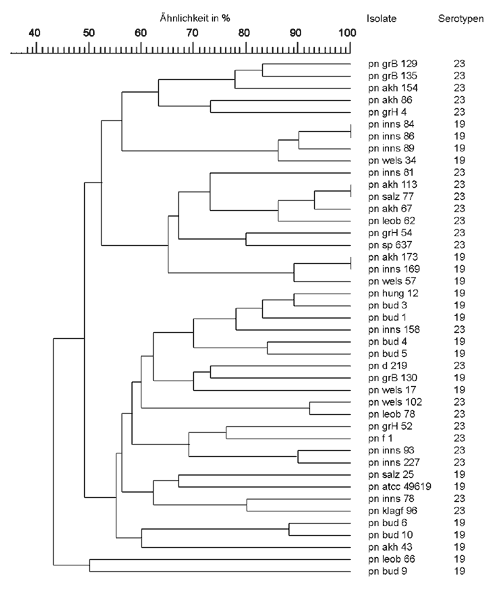
Mittels AP-PCR wurden DNA-Profile mit 4 bis 23 Banden produziert. Alle Isolate hatten eine intensive Bande bei 500 bp. Mit dieser Methode konnten vier Cluster mit je zwei bis vier Stämmen gefunden werden. Ein Cluster wurde aus vier ungarischen Stämmen gebildet. PSSP-Stämme hatten generell eine höhere Anzahl an Banden (11 bis 23) und formten zwei Gruppen: fünf Isolate mit einer Ähnlichkeit von 90% und vier Isolate mit einer Ähnlichkeit von 80%. Bei Verwendung der rep-PCR hatten die entstandenen Muster zwischen drei und 14 Banden mit einer intensiven Bande bei 500 bp. Hingegen wurden bei den ungarischen Stämmen fünf verschiedene Cluster gefunden, die aus je zwei bis fünf Stämmen bestanden (Abbildung 2). Mit den drei obengenannten molekularbiologischen Methoden konnten drei Gruppen von Stämmen aufgezeigt werden, die eine genetische Verwandtschaft besitzen.
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Im Gegensatz zu anderen europäischen Ländern, wie z. B. Frankreich, Ungarn oder Spanien [5, 8], ist das Vorkommen von PRSP in Österreich noch relativ gering, ähnlich wie in Deutschland [22], Italien [17] oder den Niederlanden [11]. Die genetische Vielfalt von S. pneumoniae-Isolaten desselben Serotypes wurde mittels verschiedener molekularer Techniken gezeigt, aber der Grad der Diversität innerhalb der Serotypen bleibt unklar [15]. Oftmals wird in der Literatur berichtet, dass die genetische Ungleichheit bei PSSP größer ist als bei PRSP [9]. Um eine eventuelle Verwandtschaft zwischen österreichischen und europäischen PRSP aufzuzeigen, wurden auch 12 Penicillin-resistente Stämme aus Ungarn, Deutschland, Frankreich und Spanien mittels verschiedener Techniken untersucht. Die verschiedensten Studien haben sich mit den unterschiedlichen Methoden zur Typisierung von Pneumokokken auseinander gesetzt [7, 10, 21]. In dieser Studie zeigte sich, dass die Auflösungskraft der einzelnen Methoden stark variiert. Unsere Ergebnisse zeigen, dass die PFGE der AP-PCR und der rep-PCR überlegen ist. Isolate, die nur intermediär empfindlich oder gegenüber Penicillin resistent waren, hatten den Serotyp 19, 19A, 23, 23B und 23F. Die PFGE zeigte, dass die Mehrheit der österreichischen PRSP vom Serotyp 23F genetisch mit dem spanischen 23F-Klon verwandt sind und in ganz Österreich zu finden sind. Auch mit dem 23F-Klon aus Frankreich zeigten einige PRSP-Isolate große Ähnlichkeit. Interessanterweise zeigte der deutsche 23F-Klon keine Verwandtschaft mit den 23- bzw. 23F-Isolaten, aber es fanden sich Gemeinsamkeiten mit zwei Pneumokokken des Serotypes 19 (ein sensitiver und ein intermediär-sensibler Stamm). Die Gründe für das Auffinden von Stämmen mit unterschiedlichen Serotypen, aber ähnlichen genetischen Ursprungs sind mannigfaltig [2, 4, 11]. Der Vergleich der Dendogramme von PFGE, AP-PCR und rep-PCR zeigt, dass alle Isolate vom Serotyp 23 ein ähnliches Muster aufweisen. Größere Unterschiede fand man bei Stämmen des Serotypes 19. Vier von sieben PRSP zeigten eine Clusterbildung, während die anderen im Dendogramm „verstreut“ waren. Weder der österreichische 19- oder 19A-PRSP zeigte eine Verwandtschaft zu den ungarischen 19- bzw. 19A-Isolaten. Das könnte auf die Existenz eines österreichischen Klons vom Serotyp 19 hinweisen. Bei der Untersuchung der PSSP fiel die große genetische Vielfalt auf. Die gefundenen Cluster bestanden nur aus jeweils zwei Stämmen und mittels keiner der angewendeten Methoden konnte eine Verwandtschaft zwischen den Penicillin-sensitiven Pneumokokken vom Serotyp 19 oder Serotyp 23 gefunden werden. Offen bleibt die Frage, warum der österreichische Serotyp 19-Klon keine Verwandtschaft mit dem ungarischen Serotyp 19 bzw. 19A-Klon zeigt, obwohl diese Länder aneinander grenzen. Im Gegensatz dazu zeigen die österreichischen PRSP vom Serotyp 23 eine große Ähnlichkeit mit dem spanischen 23F-Klon. Im Zuge der steigenden Resistenz von Pneumokokken werden Studien, die sich mit der molekularen Epidemiologie von S. pneumoniae befassen, immer wichtiger, da nur so eine klonale Ausbreitung rechtzeitig erkannt und verhindert werden kann. |
| Literatur:
1. Appelbaum P.C.: „Antimicrobial
resistance in Streptococcus pneumoniae: an overview.“ Clin. Infect.
Dis. 15 (1992) 77-83. |
| Anschrift
des Verfassers: Univ.-Prof. DDr. A. Georgopoulos Univ.-Klinik für Innere Medizin I, Abt. für Infektionen und Chemotherapie A-1090 Wien, Währinger Gürtel 18-20 |