| Pulsfeld-Gel-Elektrophorese, molekulare Analyse zur Typisierung von Mikroorganismen |
| P.E. Mrass, R. Gattringer,
W. Graninger, A. Georgopoulos Universitätsklinik für Innere Medizin I, Abteilung für Infektionen und Chemotherapie, AKH Wien (Leiter: Univ.-Prof. DDr. W. Graninger) |
| Schlüsselwörter:
PFGE, Streptococcus pneumoniae |
Zusammenfassung Typisierungsverfahren stellen eine wichtige Methode im mikrobiologischen Labor dar, um unterschiedliche Stämme eines bestimmten Bakteriengenus zu unterscheiden. Genotypische Verfahren untersuchen die Ähnlichkeit des Genoms verschiedener Isolate und können so identische, nahe verwandte, oder nicht in Beziehung stehende Stämme identifizieren. Mit ihr konnte viel über die Epidemiologie von Penicillin-resistenten Pneumokokken in Erfahrung gebracht werden. Im Moment deuten viele Publikationen darauf hin, dass die meisten Penicillin-resistenten Pneumokokken nicht von einer gemeinsamen Vorläuferzelle abstammen, dass allerdings regelmäßig Cluster klonalen Ursprungs auftreten, die sich auch über Landesgrenzen hinaus verbreiten können. |
Key-words: PFGE, streptococcus pneumoniae |
Summary Typing procedures are an important technique in the microbiologic laboratory, to discriminate different strains of a specific bacterial genus. Genotypic methods examine the similarity of the genome of different isolates, and can find identic, related and non-related strains. Pulsed-field-gel-electrophoresis (PFGE) is predominantly and because of its unique discriminatory power a very important procedure. With PFGE much could be learned about the epidemiology of penicillin-resistent pneumococci. At the moment, many papers indicate that most penicillin-resistent pneumococci do not descend from a common ancestor, still commonly clonally derived clusters appear that can spread even beyond national borders. |
|
Vor der Einführung der PFGE durch Schwartz und Cantor im Jahr 1984 [10] war eine Auftrennung von DNA-Fragmenten nur bis zu einer Größe von 50 kB möglich. Größere Fragmente zeigen bei der konventionellen Agarose-Gel-Elektrophorese eine gleiche Wanderungsgeschwindigkeit und können daher nicht aufgetrennt werden. Daher war es nicht möglich, das gesamte Genom von Bakterien, das ca. 4,5 Millionen Megabasen (4500 kB) umfasst, aufzutrennen, sondern nur winzige Bruchstücke davon. Weiters war man nicht in der Lage, intakte DNA eines vollständigen Chromosoms zu verarbeiten, da sie bei den Verdauungsschritten in den flüssigen Verdauungspuffern aufgrund der Scherkräfte, die auf die langen DNA-Moleküle wirken, in unzählige kleine Fragmente zerfallen. Es bedurfte der immensen Erfindungsgabe von Schwartz und Cantor, diese Probleme zu lösen. Das Problem der auftretenden Scherkräfte wurde bewältigt, indem intakte Bakterien in Gelblöckchen (Inserts) eingegossen wurden und alle Verdauungsschritte in diesen durchgeführt wurden. In die Inserts eingebettet, ist die DNA vor Scherkräften geschützt. Die Auftrennung von DNA-Fragmenten von einer Länge von 50 - 2000 kB wurde durch die Anwendung von gepulsten elektrischen Feldern anstatt der konventionellen homogenen Felder erreicht. Gepulste Felder sind elektrische Felder, die ihre Orientierung regelmäßig nach einem bestimmten Intervall (Pulszeit) verändern. Die DNA-Fragmente werden nach jedem Wechsel des Feldes neu orientiert und können erst dann ihre Wanderung fortsetzen. Kürzere Fragmente erreichen die neue Orientierung schneller als lange und wandern daher schneller, wodurch es zu einer Auftrennung kommt. Durch Variation der Pulszeiten kann das Optimum, bei welcher Fragmentgröße die beste Auftrennung stattfindet, modifiziert werden. Um bei Bakterien, die nur ein Chromosom enthalten, verschiedene Fragmente zu gewinnen, ist es notwendig, ein intaktes Chromosom mittels einer Restriktionsendonuklease in verschiedene Bruchstücke zu zerlegen. So erlangt man zwischen 5 und 20 Fragmente, die nach der Wanderung im Gel, Färbung in Ethidiumbromid und UV-Durchleuchtung als Banden sichtbar werden. Durch die unterschiedlichen Bandenmuster, die so entstehen, den Restriktionsfragmentpolymorphismus (RFLP), können verschiedene Bakterienstämme mit einer extrem hohen Unterscheidungskraft unterschieden werden. Um die extrem hohe Qualität dieser Methode zu unterstreichen, wird hier die Tabelle aus dem Manual of Clinical Microbiology angeführt (Tabelle 2), wo die PFGE mit anderen genotypischen Typisierungsmethoden verglichen wird, ohne auf diese genauer einzugehen. PFGE als bevorzugte Typisierungsmethode wird für die folgenden Erreger-Spezies vorgeschlagen [1]: Staphylococcus aureus, Koagulase-negative Staphylokokken, Streptococcus pneumoniae, Enterokokken, Escherichia coli (E. coli O157:H7 wird durch Serotypisieren bestimmt), Citrobacter, Proteus, Providencia, Klebsiella, Enterobacter, Serratia und Pseudomonas aeruginosa.
|
|
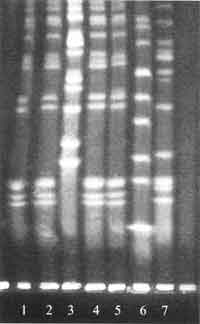
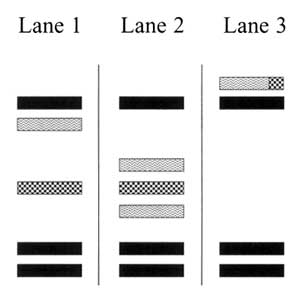
Wie schon oben darauf hingewiesen wurde, lässt sich der Verwandtschaftsgrad verschiedener Bakterienstämme durch den Vergleich der Bandenmuster, die durch ein genotypisches Typisierungsverfahren wie z.B. der PFGE geliefert werden, erkennen. Abbildung 1 zeigt die Bandenmuster von 7 Isolaten Penicillin-resistenter Pneumokokken. Lanes 1, 2 und 4 weisen identische Bandenmuster auf. Lane 5 unterscheidet sich von diesen Mustern nur durch eine Bande. Die Muster von den Lanes 3, 6 und 7 unterscheiden sich davon vollständig. Identische Stämme haben identische Bandenmuster. Aufgrund von spontanen Mutationen ist es möglich, dass sich das Bandenmuster der Nachkommen von dem ihres Ursprungsstammes unterscheidet. Abbildung 2 zeigt in Lane 1 schematisch das Bandenmuster eines Ursprungsstammes.
In Lane 2 ist das Bandenmuster zu sehen, das durch das Auftauchen eines zusätzlichen Restriktionslokus entsteht. Ein langes Fragment verschwindet, zwei kurze Fragmente treten stattdessen auf (gewelltes Muster). In Lane 3 ist durch spontane Mutation ein Restriktionslokus verschwunden. Daher entsteht ein neues langes Fragment, auf Kosten von zwei kurzen Fragmenten (kariertes Muster und gewelltes Muster). Jeweils ist durch eine einzelne Mutation eine Bandendifferenz zwischen Ursprungs- und mutiertem Stamm von 3 Banden entstanden. Durch das Einfügen oder den Verlust von einem DNA-Abschnitt ohne Restriktionslokus in ein DNA-Fragment wird jeweils eine Bande parallel nach oben oder unten verschoben, was einer Differenz von 2 Banden entspricht. Tabelle 4 fasst die durch Mutationen verursachte Änderung der Bandenmuster zusammen. Die Zahlen vergleichen jeweils den Unterschied zwischen Ursprungs- und mutiertem Stamm.
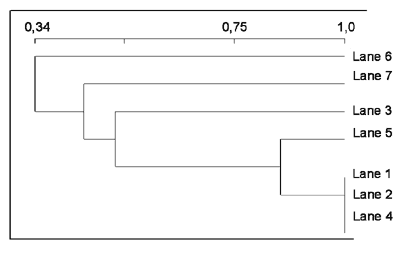
Hilfreich beim Vergleich verschiedener Bandenmuster sind Stammbäume, mit denen auf einen Blick ersichtlich wird, welche Stämme nahe verwandt oder identisch sind, bzw. welche Stämme nur sehr geringe Ähnlichkeiten aufweisen. Stammbäume werden computerunterstützt erstellt und verglichen, was einen Vergleich einer großen Anzahl von Proben in relativ kurzer Zeit ermöglicht. Abbildung 3 zeigt einen Stammbaum, der auf Basis der Bandenmuster der Lanes 1 bis 8 von Abbildung 1 erstellt wurde.
Als Faustregel bei der Analyse solcher Stammbäume kann man davon ausgehen, dass Stämme mit Bandenmustern einer relativen Ähnlichkeit von mehr als 0,8 verwandte Isolate darstellen, während Isolate mit Bandenmustern von weniger als 0,5 Übereinstimmung epidemiologisch in keinerlei Beziehung stehen. In Abbildung 3 sind daher die Pneumokokken der Lanes 1, 2, 4 und 5 verwandt, d.h. sie sind klonalen Ursprungs. |
|
Die Anwendung von genotypischen Typisierungsmethoden, insbesondere der PFGE, konnten einiges zur Aufklärung der Epidemiologie von Penicillin-resistenten Streptococcus pneumoniae beitragen. In den späten 80-er Jahren traten in Island gehäuft multi-resistente Pneumokokken auf. Interessanterweise gehörten fast alle von ihnen zum Serotyp 6B. Die Vermutung, dass es sich dabei um einen Klon handeln könnte, lag nahe. Im Jahr 1993 konnte Soares [9] durch Anwendung der PFGE auch tatsächlich zeigen, dass alle Isolate identische Bandenmuster aufwiesen. Ein multi-resistenter Klon war gefunden worden. Weiters konnte gezeigt werden, dass das Bandenmuster der Isolate dem von in Spanien isolierten multi-resistenten Pneumokokken entsprach. Vermutlich war der Pneumokokkus aus Spanien eingeschleppt worden, bevor er sich in Island ausbreitete. Weiters erschien eine Reihe von Publikationen, die als Hauptmechanismus der Ausbreitung von multi-resistenten Pneumokokken die klonale Verbreitung vermutete [5, 6, 7]. Allerdings basierte die Mehrzahl dieser Arbeiten auf der Methode der Multi-Lokus-Enzym-Elektrophorese, die nach Lefevre [4] auch für manche genetisch nicht in Verbindung stehende Isolate identische Ergebnisse liefern kann. Daher ist die Aussagekraft dieser Arbeiten nicht absolut. Im Jahr 1996 untersuchte Agnes Ferroni [2] landesweit in Frankreich Penicillin-resistente Pneumokokken mit PFGE und konnte beeindruckend die im Vergleich zu Penicillin-sensiblen Pneumokokken hohe Verwandtschaft zwischen den Isolaten zeigen. Im selben Jahr analysierte Lucinda M. C. Hall [3] im Vereinigten Königreich vorkommende Pneumokokken. Auch sie konnte Cluster von nahe verwandten Pneumokokken finden. Allerdings zeigte ein Großteil der Penicillin-resistenten Pneumokokken keinen Hinweis auf eine Abstammung von einem gemeinsamen Vorläuferstamm. Karen M. Rudolph konnte in einer Arbeit aus dem Jahr 1998 in Alaska einen Penicillin-resistenten Klon nachweisen [8], die Mehrzahl der Penicillin-resistenten Pneumokokken ließ allerdings auch in ihrer Arbeit keine Verwandtschaft erkennen. Zusammenfassend lässt sich sagen, dass die PFGE entscheidend zum momentanen Wissensstand über die Epidemiologie von Penicillin-resistenten Pneumokokken beigetragen hat. Es wurde mit Hilfe dieser Methode nachgewiesen, dass die klonale Ausbreitung von Penicillin-resisten- ten Pneumokokken tatsächlich vorkommt, ja sogar über so große Distanzen wie zwischen Spanien und Island stattfinden kann, allerdings scheinen andere Mechanismen wie horizontale Ausbreitung von Resistenzgenen oder spontane Mutation eine entscheidendere Rolle zu spielen. |
| Literatur:
1. Balows A., Hauser W.J.,
Hermann K.L., Isenberg H.D., Shadony H.J.: „Manual of Clinical Microbiology,
7th Edition.“ |
| Anschrift
des Verfassers: Univ.-Prof. DDr. A. Georgopoulos Univ.-Klinik für Innere Medizin I, Abt. für Infektionen und Chemotherapie A-1090 Wien, Währinger Gürtel 18-20 |